Imaging
Team:
Dr. N. Wagner nicole.wagner@uni-wuerzburg.de
Prof. E. Asan esther.asan@uni-wuerzburg.de
Prof. S. Ergün sueleyman.erguen@uni-wuerzburg.de
Prof. S. Karnati srikanth.karnati@uni-wuerzburg.de
Die Arbeitsgruppe-Imaging des Institutes stellt Geräte, Methoden und morphologische Expertise zur hochauflösenden mikroskopischen Analyse von Geweben und Zellen bereit. Zur Verfügung stehen ein Transmissions-Elektronenmikroskop (TEM; LEO912AB) mit digitaler Kameraeinrichtung sowie ein Konfokales System von Nikon (inverses Forschungsmikroskop Nikon Eclipse Ti), das die Möglichkeit der Probenanalyse mittels Stochastic Optical Reconstruction Microscopy (N-STORM) oder Total Internal Reflection Fluorescence Microscopy (TIRFM) bietet.
Unter anderem können folgende Untersuchungen durchgeführt werden:
- Klassische Ultrastrukturanalysen (qualitativ und quantitativ)
- Korrelative Licht- und Elektronenmikroskopie, Pre- und Postembedding Immunelektronenmikroskopie
- Konfokale Mehrkanal-Laserscanning-Mikroskopie von fluoreszenz-immunhistochemisch detektierten Proteinen oder fluorophor-markierten Proteinen in Gewebeschnitten und Zellkulturen
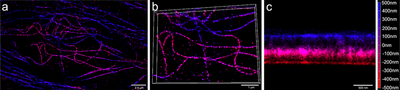
- N-STORM und 3D N-STORM zur super-hochauflösenden (laterale Auflösung bis zu 20 nm, axiale Auflösung bis zu 50nm) Lokalisation fluoreszierender Moleküle einschließlich 3-dimensionaler Rekonstruktion
- TIRFM zur selektiven Visualisierung von Fluorophoren in einer dünnen Schicht an der Probenoberfläche
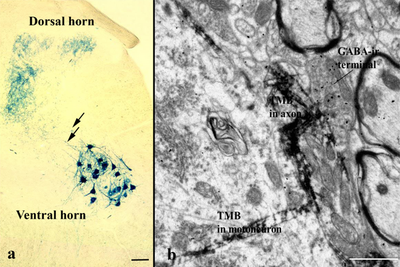
Abb. 1: Korrelative licht- (a) und elektronenmikroskopische (b) Detektion von sensorischen Axonen und Motoneuronen im lumbalen Rückenmark nach Injektion eines Meerrettich-peroxidase-markierten Tracers in den N. ischiadicus (Chromogen: Tetramethylbenzidin, TMB). Zusätzliche Postembedding-Immunogoldmarkierung zeigt GABAergen Kontakt am TMB-markierten sensorischen Axon (b). Ratte, Maßbalken in (a) 50 µm, in (b) 1 µm.
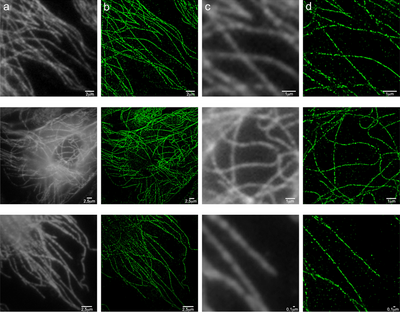
Abb.2: Vergleich zwischen konventioneller Fluoreszenzmikroskopie (a, c) und N-STORM (b, d) anhand von Mikrotubuli einer endothelialen MyEND-Zelle. Abbildungen (c) und (d) zeigen Auschnittsvergrößerungen der Aufnahmen in a und b. In aufeinanderfolgenden Aufnahmen wird die Position stochastisch angeregter Fluorophore (hier Alexa647) mit hoher Genauigkeit bestimmt und durch die Überlagerung aller Einzelbilder zu einem Gesamtbild zusammengesetzt.