Zellbiologie von Peroxisomen
Team:
Prof. S. Karnati srikanth.karnati@uni-wuerzburg.de
Dr. S. Hübner stefan.huebner@uni-wuerzburg.de
Lisa Berberich lisa.berberich@uni-wuerzburg.de
Prof. S. Ergün sueleyman.erguen@uni-wuerzburg.de
Peroxisomen sind einzelne membrangebundene, allgegenwärtige Organellen, die in allen eukaryotischen Zellen vorhanden sind. Diese Organellen sind hochdynamisch und können ihre Größe, Anzahl, den Enzymgehalt und die Proteinhäufigkeit in Abhängigkeit vom Zelltyp und den Organfunktionen oder als Reaktion auf einen metabolischen Stimulus rasch modulieren. Bisher sind mehr als 130 verschiedene peroxisomale Proteine an einer Vielzahl von Stoffwechselwegen beteiligt, wie der Synthese von Cholesterin und Plasmalogenen, dem Abbau verschiedener toxischer, bioaktiver entzündungsfördernder und signalgebender Lipidderivate und dem Metabolismus reaktiver Sauerstoffspezies. Peroxisomale Dysfunktion wurde mit schweren Stoffwechselstörungen in Verbindung gebracht, und diese subzellulären Organellen gelten heute als schützende Organellen mit größerer Bedeutung für die menschliche Gesundheit und potenziellen Auswirkungen auf verheerende Erkrankungen des Menschen. Die molekularen Funktionen und die Beteiligung von Peroxisomen an Herz-Kreislauf-Erkrankungen (CVD) sind jedoch weiterhin unbekannt. Ziel unserer Arbeitsgruppe ist es daher, „die peroxisomabhängigen molekularen Mechanismen und zellspezifischen-peroxisomalen Funktionen bei der Pathogenese von Herz-Kreislauf-Erkrankungen zu identifizieren und neue Therapiestrategien durch Modulation des Peroxisomsignals zu entwickeln. Um diese Ziele zu erreichen, verwenden wir In-vitro-, In-vivo-, Ex-vivo-Modelle und hochauflösende mikroskopische (SIM, STORM) sowie ultrastrukturelle Techniken (TEM, REM und ImmunoTEM), um molekulare Funktionen und die Beteiligung von Peroxisomen an CVDs zu untersuchen.
Der Fokus der AG-Peroxisome ist hauptsächlich interessiert an:
- Bildgebung der Peroxisome
- Aufklärung der funktionellen Rolle von Peroxisomen bei der Entzündung und Regeneration des Herzens
- Etablierung von Herz-spezifischen peroxisomalen Knockout-Maus-Modellen
Im Speziellen gehen wir folgenden Fragestellungen nach:
- Wie sind die peroxisomalen Docking-Komplex-Proteine PEX13p und PEX14p in der peroxisomalen Membran aufgebaut?
- Gibt es eine Beziehung zwischen der Häufigkeit der Peroxisomenproteine und der Größe und Form des Peroxisoms?
- Ob und welche strukturellen Veränderungen der peroxisomalen Membranproteinen bei pathologischen Prozessen eine Rolle spielen?
- Wie und warum interagieren Peroxisomen mit Mitochondrien oder mit anderen Zellorganellen?
- Welche Rolle spielen Peroxisomen und die damit verbundenen Signale in den vaskulären Endothelzellen und wie werden hierbei die Prozesse der Atherosklerose beeinflusst? Über welche Mechanismen werden dabei die peroxisomalen Gene reguliert?
- Welche peroxisomalen Enzyme kommen in Megakaryozyten und Thrombozyten vor? Spielen Peroxisomen eine Rolle bei deren Differenzierung dieser Zellen bzw. ihrer Derivate?
- Welche Lipide / Metabolite, die ausschließlich von Peroxisomen abgebaut werden, sind an den Entzündung- und/oder Regenerationsprozessen des Herzens beteiligt? Ob das veränderte Lipidprofil und die damit einhergehenden funktionellen Veränderungen bei diesen Vorgängen durch die PPAR-Peroxisomenschleife vermittelt werden?
- Welche Rolle spielt der peroxisomale ROS-Stoffwechsel bei der Pathogenese von Herz-Kreislauf-Erkrankungen?
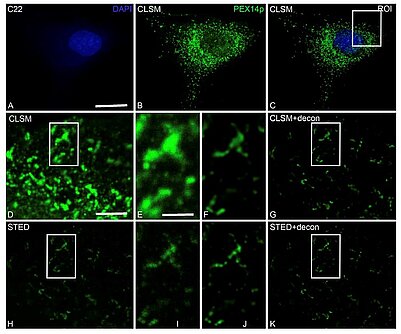
Abb. 1: C22-Zellen wurden auf PEX14p angefärbt und mit CLSM und STED abgebildet, gefolgt von Dekonvolution (A-K). Der interessierende Bereich des CLSM-Bildes (C) ist mit niedriger Vergrößerung (D), hoher Vergrößerung (E), CLSM-niedriger Vergrößerung mit Dekonvolution (G) und hoher Vergrößerung (F) gezeigt. Die gleichen interessierenden Bereiche wurden einer STED-Bildgebung mit geringer Vergrößerung (H), starker Vergrößerung (I), STED-geringer Vergrößerung mit Dekonvolution (K) und starker Vergrößerung (J) unterzogen. Bars: a-c: 15 μm; d, g, h, k: 5 um; e, f, i, j: 1 um.
Abb. 2: C22-Zellen wurden auf PEX14p angefärbt und mit STORM abgebildet (A-C). Ansicht einer Zelle (A) mit geringer Vergrößerung, gefolgt von einer höheren Vergrößerung von zwei ausgewählten Peroxisomen (B, C).
Ausgewählte Publikationen:
- Florian Kleefeldt, Heike Bömmel, Britta Broede, Michael Thomsen, Verena Pfeiffer, Philipp Wörsdörfer, Srikanth Karnati, Nicole Wagner, Uwe Rueckschloss, Süleyman Ergün. Aging-related carcinoembryonic antigen-related cell adhesion molecule 1 signaling promotes vascular dysfunction
Aging Cell. 2019;18(6):e13025. doi:10.1111/acel.13025
- Karnati S*, Oruqaj G, Janga H, Tumpara S, Colasante C, Van Veldhoven PP, Braverman N, Pilatz A, Mariani TJ, Baumgart-Vogt E (2018) PPARα-mediated peroxisome induction compensates PPARγ-deficiency in bronchiolar club cells.
PLoS One. 2018 Sep 13;13(9):e0203466. doi: 10.1371/journal.pone.0203466.
- El-Merhie N, Baumgart-Vogt E*, Pilatz A, Pfreimer S, Pfeifer B, Pak O, Kosanovic D, Seimetz M, Schermuly RT, Weismann N, Karnati S* (2017) Differential alterations of the mitochondrial compartment and respiratory chain complexes during postnatal development of the mouse lung
Oxidative medicine and cellular longevity. 1.Volume 2017, Article ID 9169146, https://doi.org/10.1155/2017/9169146
- Vijayan V, Tumpara S, Karnati S, Kamlayan L, Immenschuh S, Reddy S, Linke M, Weichhart T, Schmid T, Baumgart-Vogt E (2017)
A New Immunomodulatory Role for Peroxisomes in Macrophages Activated by the TLR4 Ligand Lipopolysaccharide.
Journal of Immunology, Mar 15;198(6):2414-2425 - Zaki MS, Heller R, Thoenes M, Nürnberg G, Stern-Schneider G, Nürnberg P, Karnati S, Swan D, Fateen E, Nagel-Wolfrum K, Mostafa MI, Thiele H, Wolfrum U, Baumgart-Vogt E, Bolz HJ (2016) PEX6 is expressed in photoreceptor cilia and mutated in deafblindness with enamel dysplasia and microcephaly
Hum Mutat. Feb;37(2):170-4.
- Oruqaj G, Karnati S, Vijayan V, Kotarkonda LK, Boateng E, Zhang W, Ruppert C, Günther A, Shi W, Baumgart-Vogt E (2015) Compromised peroxisomes in idiopathic pulmonary fibrosis, a vicious cycle inducing a higher fibrotic response via TGF-β signaling.
Proc Natl Acad Sci. 112(16):E2048-57. (§ equal contribution)
- Karnati S, Palaniswamy S, Alam MR, Oruqaj G, Stamme C, Baumgart-Vogt E (2015) C22-bronchial and T7-alveolar epithelial cell lines of the immortomouse are excellent murine cell culture model systems to study pulmonary peroxisome biology and metabolism
Histochemistry and Cell Biology. Mar;145(3):287-304
- Karnati S, Lüers G, Pfreimer S and Baumgart-Vogt E (2013)
Mammalian SOD2 is exclusively located in mitochondria and not present in peroxisomes.
Histochemistry and Cell Biology, Aug:140(2):105-17 - Karnati S, Baumgart-Vogt E (2009) Peroxisomes in airway epithelia and future prospects of these organelles for pulmonary cell biology
Histochem Cell Biol. Apr: 131(4):447-54.
- Karnati S, Baumgart-Vogt E (2008) Peroxisomes in mouse and human lung: Their involvement in pulmonary lipid metabolism.
Histochem Cell Biol. Oct; 130(4):719-40